在過去的數年中,全球公共衛生安全面臨嚴峻挑戰。尤其自新冠疫情爆發以來,其病原體——嚴重急性呼吸綜合征冠狀病毒 2 型(SARS-CoV-2)持續進化,并相繼出現多個主導疫情走勢的變異株,它們具有不同的感染能力和逃避免疫應答能力,極大增加了疫情防控工作難度和醫療系統負擔。
公共衛生檢測和 SARS-CoV-2 基因組測序是全面檢測流行變體的重要手段,但此類臨床監測往往高度依賴大量實驗室資源,且需個體主動參與檢測,難以全面追蹤 SARS-CoV-2 變異株出現和傳播。尤其在醫療資源相對較少或檢測意愿相對較低的地區,臨床監測更容易出現檢測偏差,形成防控盲區。
作為一種補充手段,利用基于廢水的流行病學(Wastewater-based epidemiology, WBE)評估社區感染情況自上世紀 40 年代提出以來,已為疾病爆發預警發揮了重要作用。WBE 主要通過分析廢水中人體排出的病毒痕跡,檢測并追蹤病毒組成和動態變化。相比臨床監測,WBE 不依賴個體主動檢測就能客觀且無偏差地反映覆蓋區域內群體感染態勢,實現提前預警,具有顯著的成本效益。
需要基于已知變體的突變模式(如 GISAID 或 UshER 數據庫中的參考序列)進行檢測,若出現未在臨床中表征或未被收錄的新型變異株時,往往難以精準識別,這在一定程度上制約了 WBE 的檢測效能。
針對于此,來自內華達大學拉斯維加斯分校的研究團隊提出一種名為 ICA-Var(Independent Component Analysis of Variants) 的多變量分析方法,該方法基于無監督機器學習流程設計,通過獨立成分分析( Independent Component Analysis, ICA)從廢水數據中提取共變和時間演變的突變模式,實現了更早、更準確的變異株檢測。
基于該方法,研究團隊在 2021 年底到 2023 年間,準確檢測到 Delta 變異株、Omicron 變異株和重組 XBB 變異株。此方法不僅再次印證了廢水監測對疫情防控早期預警的有效性,同時也為缺乏臨床監測條件下全面追蹤病毒變異和傳播提供了新工具。
相關研究以「Early detection of emerging SARS-CoV-2 Variants from wastewater through genome sequencing and machine learning」為題,發表于 Nature Communications。
研究亮點:
* 該方法揭示了城市和農村地區病毒變異的時空動態,證實病毒從城市向農村傳播的規律,為醫療可及性差的地區或缺乏臨床測序數據的情況,提供了行之有效且低成本的變異株檢測范式
* 與現行金標準工具 Freyja 相比,ICA-Var 的多變量分析方法更具顯著優勢,在對 Delta、Omicron 及最新 EG.5、HV.1、BA.2.86 等變異株的檢出時間平均提前 1-4 周

長周期、多點位數據采集
在本次研究中,實驗所采用的廢水樣本來自于 2021 年 8 月至 2023 年 11 月間,從內華達州南部的城市和農村地區收集到 3,659 份廢水樣本。采集后的廢水樣本會先置于現場冰上,冷藏保存直至處理,保存時間不超過 36 小時。
在核酸提取過程中,研究團隊先使用 Promega 公司生成的 Wizard Enviro Total Nucleic Kit(貨號 A2991),并按照規范要求從廢水樣本中分離核酸。與此同時團隊修改了 Promega 方案,用蛋白酶溶液裂解廢水,并使用 Macherey-Nagel 公司生成的 NucleoMag Beads(貨號 744970)結合游離核酸。對于總量大于 10 ng 的 RNA,研究團隊使用 New England BioLabs 公司的 LunaScript RT SuperMix Kit 進行處理,用于第一鏈 cDNA 合成。
測序文庫構建與測序方面,研究團隊使用 Paragon Genomics 公司的 CleanPlex SARS-CoV-2 FLEX Panel 構建擴增子測序文庫,隨后文庫在 Illumina NextSeq 500 或 NextSeq 1000 平臺上,使用 300 循環流動槽進行測序。
測序數據的處理方面,團隊先用 cutadapt 軟件(4.2 版本)去除測序讀段對中的 Illumina 接頭序列。然后用 bwa mem 軟件(0.7.17-r1188 版本)將測序讀段對映射到 SARS-CoV-2 參考基因組(NC_045512.2)。接著用 fgbio TrimPrimers 工具(2.1.0 版本,硬剪切模式)去除比對讀數中 Paragon Genomics 的 CleanPlex SARS-CoV-2 FLEX 擴增子引物序列。最后用 iVar variants 軟件(v1.4.1 版本)檢測變異位點(基于與 2020 年初始參考基因組的等位基因頻率差異),并通過 samtools 軟件(v1.16.1 版本)計算基因組覆蓋率和讀取深度。
剩余 2,684 份樣本用于質量控制(Quality Control, QC)分析。之后通過嚴格的質量控制,僅保留測序深度達到 50 倍且覆蓋 SARS-CoV-2 基因組 80% 以上的廢水樣本用于后續分析。如下圖所示:
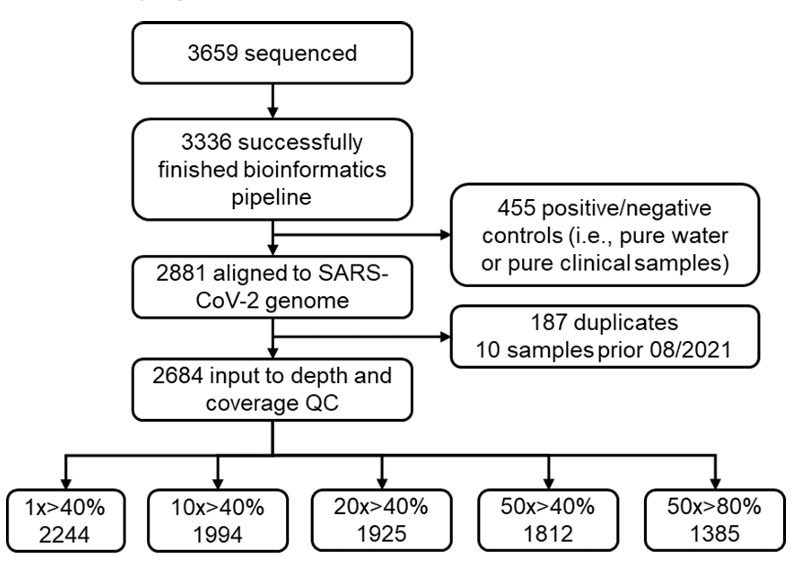
廢水樣本的詳細篩選流程
最終,研究采用了 1,385 個高質量樣本,涵蓋 SARS-CoV-2 變異株的 59,422 個突變位點,用于后續分析。
為輔助驗證 ICA-Var 方法的有效性,研究團隊使用臨床數據作為對照和參考依據,分析了從 GISAID 數據庫下載的 8,810 個內華達州具有高覆蓋度的臨床 SARS-CoV-2 序列數據,時間覆蓋 2021 年 9 月至 2023 年 11 月。
以 ICA 為核心,引入雙回歸方法打造新冠病毒檢測新工具
ICA-Var 的核心流程,是通過獨立成分分析處理廢水樣本中的突變頻率,提取獨立的共變異突變模式,然后通過雙回歸方法(dual-regression)將這些模式與原始樣本關聯,以實現對病毒變異株的追蹤。如下圖所示:
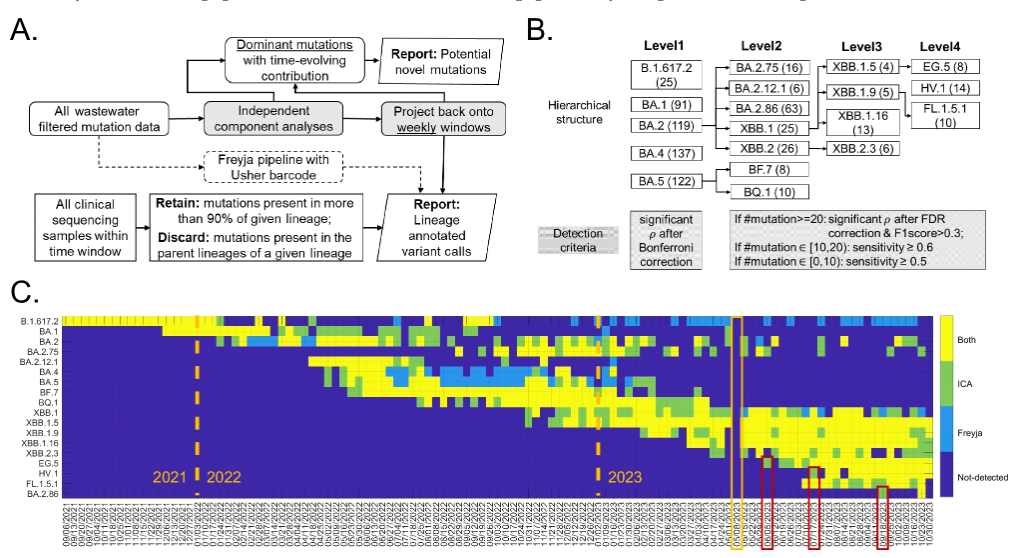
ICA-Var 方法流程,及與 Freyja 的比較
* 圖中 A 為獨立成分分析流程。兩個矩陣分別為:每周 SARS-CoV-2 譜系檢測(底行)和潛在的新突變(頂行)
* 圖中 B 為 18 種關切變異株的層次結構。每種變異株的主要突變位點(即譜系定義位點)取自 covspectrum.org 總結的臨床數據,括號內為主要突變的數量;陰影框為擬議流程中進行檢測的標準
*圖中 C 為 ICA-Var 方法與最先進工具 Freyja 的比較。對于新出現的變異株 EG.5、HV.1 和 BA.2.86,紅色框表示 ICA-Var 檢測時間更早;黃色框表示由于技術問題未進行廢水采樣的一周。
具體來說,由于廢水樣本中的 SARS-CoV-2 基因組信號是多種變異株混合的結果,且受到樣本降解、測序誤差等干擾,傳統方法難以直接解析單一變異株的特征。為此,ICA-Var 的核心思路是利用獨立成分分析——這一盲源分離技術,假設混合的突變信號是有多個「獨立來源」線性組合而成,通過數學建模將這些獨立模式從混合數據中拆解出來。
研究團隊首先對數據進行了預處理,通過對廢水樣本的 SARS-CoV-2 基因組測序數據進行質量控制,過濾低質量讀段和噪聲突變,構建「突變頻率矩陣」,用行代表樣本,列代表突變位點,值為該位點在樣本中的突變頻率。之后對突變頻率矩陣進行獨立成分分析,將混合信號分解為獨立成分,每個成分代表一組「共變異突變模式」,即某一變異株的特征性突變組合,這些突變在樣本中隨時間同步出現或消失。
此間,研究利用最小描述長度(Minimum Description Length, MDL)準則確定獨立成分數量,利用 fastICA 算法執行獨立成分分解。為確保結果可靠,他們用不同初始值重復 ICA 分析 50 次,借助 ICASSO 軟件對每次運行得到的成分進行聚類和可視化,最終只保留緊密聚類對應的可靠估計作為源矩陣。
之后,為進一步確定每周的變異株情況,研究團隊通過雙回歸方法,將獨立成分分析得到的源矩陣重新投射到原始樣本中,計算每個樣本中個獨立成分的「貢獻度」,即該變異株在樣本中的相對豐度,從而量化出不同變異株在時間和空間上的動態變化,如出現時間、流行趨勢、城鄉分布差異等。
研究團隊使用全樣本源矩陣作為一般線性模型(General Linear Model, GLM )中的一組源回歸量,以找到與全樣本源矩陣相關的每周樣本的信號拆解規律。然后在第二個 GLM 中,使用每周樣本的信號拆解規律作為回歸量,以找到仍與全樣本源矩陣相關的周特異性源矩陣。該過程生成構成對偶空間的估計值對,共同為每個周樣本中的原始全樣本獨立成分分析源矩陣提供最佳近似。
最后,研究團隊將分離出來的獨立成分與臨床測序數據中已知變異株進行對比并進行注釋,從而成功確定其對應的變異株,或篩選出未匹配的共變突變模式,以預警新的變異株可能性。
ICA-Var 方法克服了傳統方法依賴「預定義參考變異株條形碼」的弊端,通過捕捉突變的共變模式,實現了比傳統方法更早、更準確識別新型變異株。同時,結合了雙回歸分析,該方法還揭示了城鄉傳播差異,以及突變位點的時間進化趨勢。總而言之,ICA-Var 為新冠病毒檢測提供了一個更靈敏、更全面,且兼顧成本效益的工具。
檢測效能超越現行金標準工具 Freyja,具備預測新變異株的潛力
為了驗證和評估 ICA-Var 的性能,研究團隊將其與現行的金標準工具 Freyja 進行了對比,后者是一種用于估算廢水中存在的新冠病毒譜系相對豐度的工具,利用一個由界定譜系的突變所構成的「條形碼」文庫,來唯一確定所有已知的新冠病毒譜系,并采用深度加權、最小絕對偏差回歸方法來求解譜系豐度。實驗證實 ICA-Var 的多變量分析方法更具顯著優勢。
如下圖所示。模型方法及架構部分簡單闡述了 ICA-Var 能夠更早地檢出新變異株 EG.5、HV.1 和 BA.2.86,主要內容將在本部分將拓展來講。
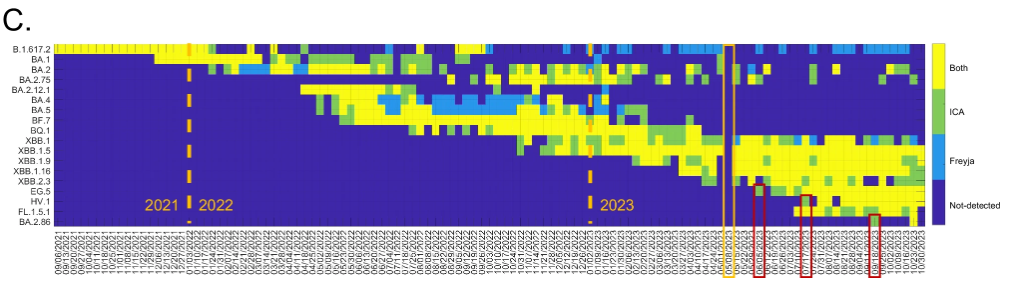
ICA-Var 與 Freyja 的比較
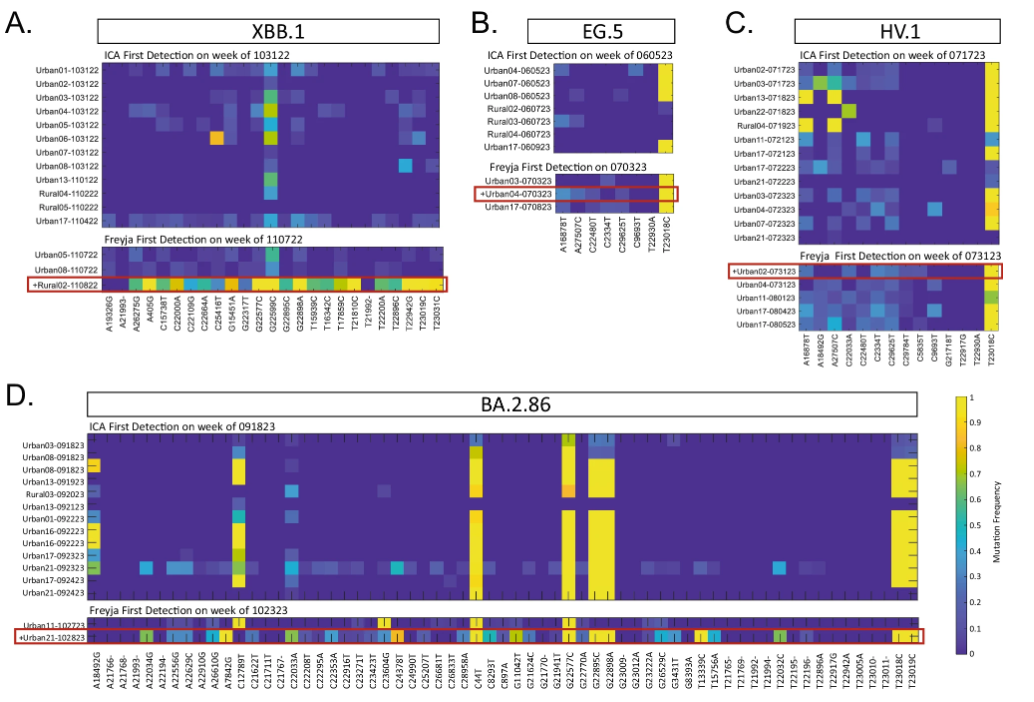
在檢測 XBB.1、EG.5、HV.1 和 BA.2.86 四種變異株時 ICA-Var 與 Freyja 的對比
具體來說,在 2022 年,ICA-Var 證明能比 Freyja 早一周或數周時間,檢測到 BA.2、BA.4、BA.5、BF.7、BQ.1、XBB.1 和 XBB.1.5 等變體。而在 EG.5 的檢測中,ICA-Var 在 6 月 5 日那一周便檢測出這種變體,但 Freyja 在 7 月 3 日才識別出 EG.5 的信號,此時廢水樣本豐度達 23.08%,在 8 個 EG.5 顯性突變位點中已有 5 個位點顯示。同樣的,對于 XBB.1、HV.1 和 BA.2.86 等變異株,ICA-Var 也比 Freyja 早幾周檢測到。
這得益于 ICA-Var 通過整合多個樣本中那些「可靠但優勢突變位點流行率較低」的信息,提升了統計效力,從而實現了更早的檢測。這意味著它不必依賴于單個樣本中高比例的優勢突變,只需匯總多個樣本的微弱信號即可增強檢測能力;相比之下,Freyja 則要求至少有一個單獨的樣本明確出現優勢突變位點才能完成檢測,這也意味著它更依賴于單個樣本中足夠明顯的突變信號,對微弱或分散的信號并不敏感。
實驗進一步檢測了城鄉樣本中變異株的動態趨勢。研究團隊從 2022 年初開始,對內華達州南部農村地區的廢水樣本進行了測序和分析,并進行了全面的城鄉流行病學比較,每周分別對城市和農村樣本進行分析。
結果顯示,ICA-Var 和 Freyja 在 18 種關切變異株中,均先在城市廢水樣本中檢測出 16 種 SARS-CoV-2 變異株,之后才在農村樣本中發現,這表明了病毒變異株通常先在城市出現,然后再擴散到農村的規律。如下圖所示:
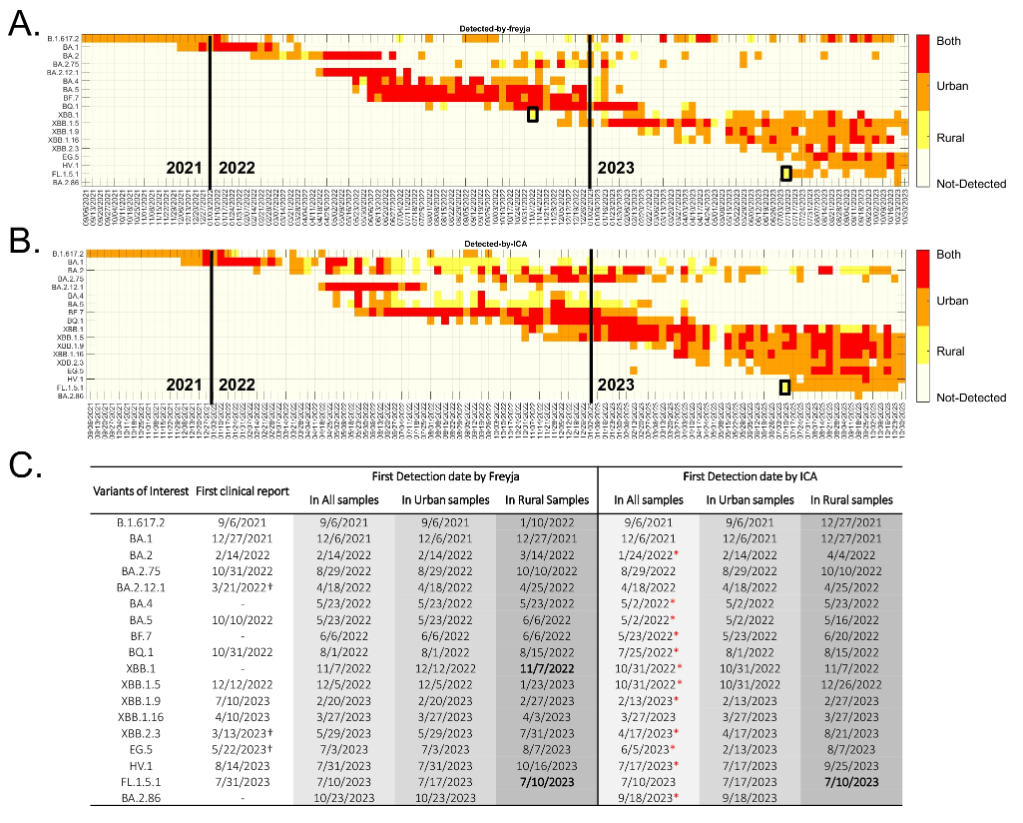
城鄉廢水樣本的變異檢測
例外的是,Freyja 最初在農村廢水樣本中檢測到了 XBB.1,而 ICA-Var 則比之早一周在城市廢水樣本中發現了該變異株;兩個工具都在農村廢水樣本中發現了 FL.1.5.1,而同期城市廢水樣本中,該變異株顯性突變的替代等位基因頻率和流行率均低得多。
研究還揭示了突變位點的時間進化趨勢。研究團隊將在 2021 年 8 月到 2023 年 11 月間 177 個「時間進化貢獻顯著的突變位點」,與 B.1.617.2、BA.1 和 XBB.1 變異體的顯性突變位點進行了比較,如下圖所示:
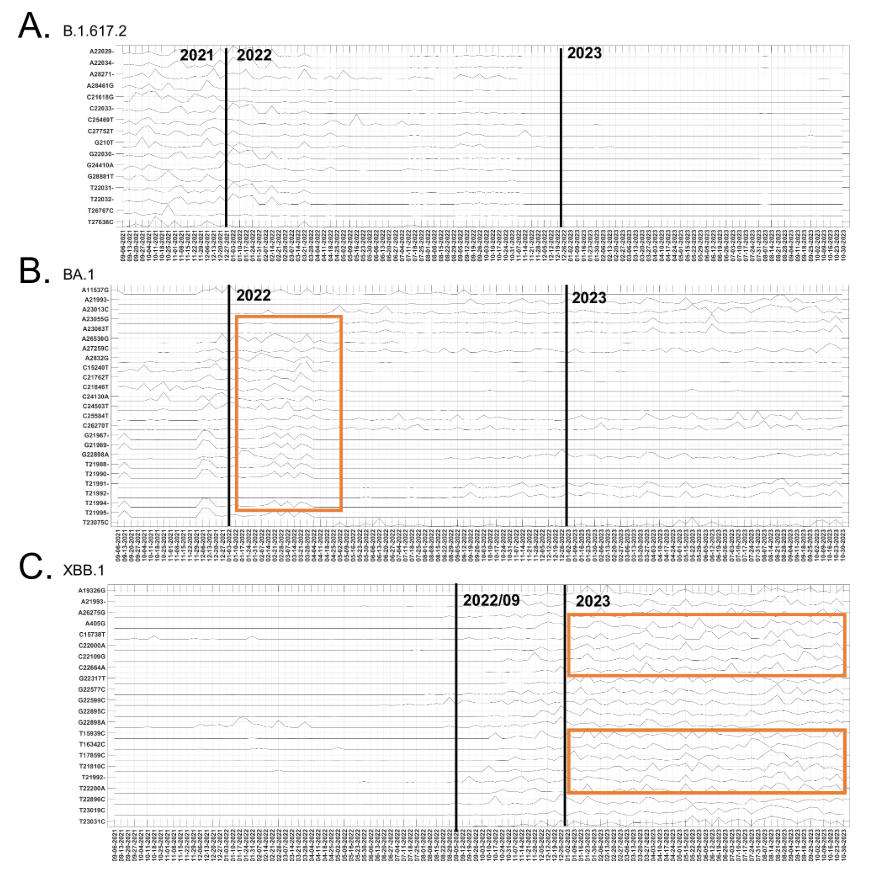
所提方法中具有顯著時間進化貢獻的突變
Delta 變異株(B.1.617.2)的 25 個主要突變位點中,16 個在 2021 年末出現顯著波動貢獻,隨后在 2022 年逐漸下降;Omicron 亞型 BA.1 的相關突變貢獻在 2021 年末明顯增加,于 2022 年初達到峰值,部分 BA.1 突變位點的貢獻在 2023 年仍有波動,且在其他 Omicron 亞譜系中被發現,如 XBB.1;XBB.1 變異株的 25 個主要突變中,22 個表現出明顯的時間動態貢獻,2022 年 9 月后影響顯著。多個突變位點呈現相似波動模式,表明它們存在共變異,體現了 XBB.1 的重組特性。
這些分析表明了 ICA-Var 鑒定的突變位點的時間進化貢獻與 Delta、Omicron 和 XBB.1 變異的臨床發現是一致的,進一步說明了 ICA-Var 結果的可靠性,證明其具有識別可能導致新變體出現的新突變模式的潛力。
實驗為此進行了詳細驗證,研究團隊通過與 15 種主要變異株的顯性突變位點交叉比較,篩選出 113 個潛在的新突變位點,然后采用層次聚類算法將這些突變位點分為 6 個特征簇。如下圖所示:
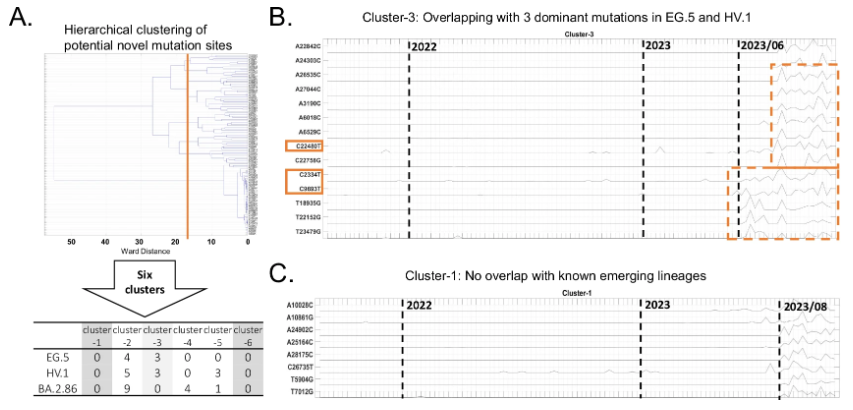
未來可能出現的突變模式
在這些特征簇中,其中 4 個(簇 2-5)的突變位點與 2023年末出現的變異株存在重疊。簇 1 和 簇 6 沒有與已知突變位點的重疊突變。其中簇 1 的突變位點在 2023 年 8 月后呈現明顯的共變異模式,對其中 8 個突變位點在 GISAID 的臨床測序數據中驗證發現,它們在臨床樣本中報告頻率較低。因此,**這些突變可能導致新型新冠病毒變異株的出現,有待臨床測試進一步驗證,**需要密切監測。
機器學習加持,廢水監測不斷迭代推動高質量病毒防控
正如開篇所提,WBE 并非一種新方法,早在上世紀 40 年代,環境病毒學家就認識到通過廢水中細胞培養試驗獲得脊髓灰質炎病毒的價值,此后 WBE 經過不斷改進,進而成為用于疾病爆發早期預警的有效工具。新冠疫情爆發以來,WBE 再次為疫情防控工作起到了一定積極作用。
比如在 2023 年末,就有報道提到瑞典的研究團隊通過整合污水和 COVID-19 病例的基因組檢測,成功早期檢測到新型 SARS-CoV-2 BA.2.86 變異株的出現。除此之外,為了更加有效、積極的利用 WBE 進行新冠病毒變體檢測,不少實驗室還通過開發或改進相關模型,為 WBE 提供更具成本效益的工具。
如清華大學、河北科技大學和天津生態環境監測中心的研究人員聯合發表的題為「Validation of methods for enriching and detecting SRAS-CoV-2 RNA in wastewater」的研究。研究通過對比超濾(ultrafiltration)和共價親和樹脂分離(covalent affinity resin separation)兩種濃縮技術,結合逆轉錄定量 PCR(RT-qPCR)和逆轉錄數字 PCR(RT-dPCR)兩種檢測方法,評估其在廢水病毒監測中的性能。
最后,**研究指出逆轉錄數字 PCR(RT-dPCR)方法是檢測廢水中低濃度 SARS-CoV-2 RNA 的更優選擇,**檢出率更高,且對PCR抑制物具有較好耐受性。
* 論文地址:
https://link.springer.com/article/10.1007/s10311-025-01843-6
另外,加拿大阿爾伯塔大學病理和實驗醫學系教授李杏放教授團隊發表的題為「Quantification and Differentiation of SARS-CoV-2 Variants in Wastewater for Surveillance」的研究。他們在此前為臨床樣本開發的 Gamma (ABG)和 Delta 多重 RT-qPCR 檢測方法基礎上,針對 Omicron 亞變體,利用其獨特的突變,開發了 Omicron 三重 RT-qPCR 檢測法,能區分五種主要的 Omicron 變異株亞系。這是單管 RT-qPCR 三重測定法在一年的時間內檢測和鑒定廢水樣本中的所有 Omicron 亞變體的第一項研究。
* 論文地址:https://pubs.acs.org/doi/10.1021/envhealth.3c00089
總而言之,當今世界面臨嚴峻的公共衛生安全挑戰,廢水監測作為一種高效的群體監測手段,正發揮著不可替代的作用。而隨著技術迭代,廢水監測亦將不斷發展,從早期依賴已知變異模式的靶向檢測,逐步向全基因組測序、未知病原體識別等方向突破,靈敏度與覆蓋面持續提升,為疫情預警、溯源及政策制定提供更精準的關鍵數據,成為公共衛生安全防線的重要補充。
參考資料:
1.https://www.nature.com/articles/s41467-025-61280-5
2.https://mp.weixin.qq.com/s/ZzzZt-uNNc5DsD-ib3Ww8g
3.https://mp.weixin.qq.com/s/qFQfayoNJ7ZME11GBFlqvQ
4.https://mp.weixin.qq.com/s/meuN
來源: HyperAI超神經


 科普中國公眾號
科普中國公眾號
 科普中國微博
科普中國微博

 幫助
幫助
 HyperAI超神經
HyperAI超神經 
